Chaque individu est unique… et cela complique un peu la recherche clinique. En effet, pour mesurer l’efficacité d’un médicament, on réalise des essais cliniques grâce à des volontaires. Mais cette mesure est difficile, car tout le monde ne réagit pas de la même manière à un traitement. Certains manifestent des effets clairs, quand d’autres y semblent insensibles ou y réagissent mal. Pour comprendre l’effet d’un médicament, il faut donc comprendre cette variabilité d’un individu à l’autre. C’est le défi que tente de relever Marc Lavielle, du Centre de mathématiques appliquées de l’École polytechnique et directeur de recherche à l’INRIA.
Mathématiques à l’appui, il aide les médecins et les biologistes à mieux comprendre les effets des médicaments qu’ils testent. « J’analyse des données d’essais cliniques, par exemple de phase 1 qui impliquent peu de patients. Puis avec mes collaborateurs, je modélise la pharmacocinétique du médicament. » Algorithmes et statistiques lui permettent ainsi de reproduire numériquement le devenir du médicament dans le corps. « Grâce à ce modèle, il sera possible d’optimiser la construction de la phase 2 et donc d’augmenter les chances d’obtenir un résultat significatif ». Car découvrir au bout de plusieurs semaines de travail que l’on ne sait toujours pas si un médicament est efficace ou non est l’une des grandes craintes de la recherche clinique.
Pour éviter cet écueil, Marc Lavielle propose de mieux prendre en compte la diversité des individus. Il s’agit de trouver comment on peut choisir les patients qui répondent le mieux au traitement et éviter ceux qui risquent des effets secondaires. Mais ce n’est pas facile à déterminer. Il faut comprendre quels signes biologiques ou quels gènes permettent de prédire la réponse d’un individu. Caractériser ces effets est au cœur de la démarche de médecine personnalisée : lorsque le médicament sera autorisé, les médecins ne le prescriront qu’aux personnes susceptibles d’y réagir favorablement.
Intégrer la variabilité interindividuelle
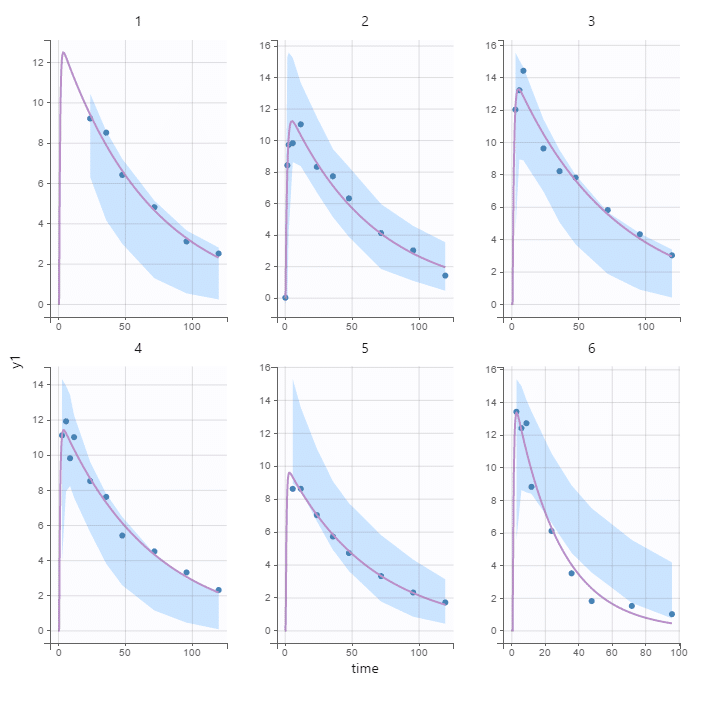
Les travaux de Marc Lavielle reposent sur ce qu’on appelle « les modèles à effet mixte », et que l’on peut représenter avec un graphique. « Tous les modèles ont la même forme. Ils commencent par la phase d’absorption du médicament, sa distribution dans l’organisme puis sa métabolisation et, enfin, son élimination. » Mais pour chaque personne étudiée, ce modèle change un peu. « Selon les individus, des paramètres varient : certaines personnes vont absorber le médicament plus lentement, d’autres vont l’éliminer plus vite, etc. »
Ces différences seront parfois expliquées par des paramètres classiques de la médecine, comme le poids du patient. Mais avec le développement des nouvelles techniques de séquençage de l’ADN, on peut désormais également identifier d’autres variables, appartenant à la génétique. « La pharmacogénétique, cherche à utiliser des informations génétiques pour comprendre pourquoi certains patients répondent à un traitement et pas d’autres », explique-t-il. « La probabilité de succès du modèle va dépendre de notre capacité à expliquer la variabilité entre les individus. »
Pour cela, le chercheur nourrit ses outils mathématiques avec des données biologiques. « Il est fondamental de travailler avec des spécialistes du domaine pour interpréter les résultats et s’attacher aux paramètres qui ont du sens d’un point de vue biologique », insiste Marc Lavielle.
Optimiser la recherche clinique
Les termes du problème sont posés, il ne reste qu’à trouver la bonne équation, celle qui décrit le devenir du médicament ou l’évolution de la tumeur au cours du temps, tout en intégrant la manière dont ces processus varient d’un individu à l’autre. Ici se niche l’art du mathématicien. Après cela, il pourra retourner présenter son outil de simulation aux médecins.
« Avec ces modèles, on peut générer des patients virtuels et simuler leur réponse », précise Marc Lavielle. On parle alors d’essais cliniques virtuels. « L’avantage des patients virtuels, c’est qu’on peut leur donner n’importe quel traitement, cela ne fera pas de mal… On s’affranchit des contraintes éthiques, et surtout on gagne du temps ! » Un modèle numérique s’exécute en quelques secondes, à comparer aux uns ou deux ans exigés en vie réelle par une phase 1 – qui étudie la tolérance au médicament et aide à définir la dose et la fréquence d’administration recommandées pour les études suivantes.
Ces modèles peuvent être appliqués à tous les essais cliniques, en médecine personnalisée ou non. « Mais lorsqu’ils sont construits à partir des données d’un essai de phase 3 [qui évalue l’efficacité du traitement], c’est-à-dire des données de milliers d’individus, on obtient une très belle description du comportement du médicament. », indique Marc Lavielle. « En combinant les informations cliniques d’un patient particulier avec celles de l’essai clinique, on sera plus tard en mesure de prévoir sa réponse au traitement. »
Prédire la réponse des patients
C’est l’opportunité d’identifier les patients susceptibles de répondre à un traitement, de manifester des effets secondaires ou de développer des résistances. Autant de questions cliniques importantes. Résultat : la recherche biomédicale manque de mathématiciens. Ce n’est donc pas un hasard si le géant biopharmaceutique Sanofi finance depuis décembre 2019 un programme de mécénat auprès de l’Institut Polytechnique sur le thème « Innovation numérique et science des données en santé ».
En parallèle, des start-ups investissent la question. Les idées de Marc Lavielle, associées à celles de Jérôme Kalifa ont ainsi donné naissance à Lixoft en 2011. L’entreprise commercialise ainsi un logiciel d’aide à la conception d’essais cliniques. Elle pourrait aussi se lancer sur le marché des dispositifs médicaux avec un outil pour identifier, à partir de leurs données biologiques, les patients les plus susceptibles de répondre ou non à un traitement. Jonathan Chauvin, son PDG, raconte : « Nous sommes encore en train de réfléchir à cette option. Mais cela implique beaucoup de contraintes réglementaires ». Un autre calcul stratégique.









